Staging automatico del cancro tramite analisi delle immagini basata su IA
ARCADIA è un’iniziativa di ricerca rivoluzionaria sviluppata in collaborazione con CRS4, il principale centro di ricerca della Sardegna. Introduce un sistema sofisticato progettato per supportare i medici nella diagnosi accurata del cancro colon-rettale. Utilizzando scansioni ecografiche 3D, ARCADIA automatizza la segmentazione del tumore, la classificazione dello stadio e identifica i linfonodi infiltrati.
Introduzione
L’automatizzazione dello staging del cancro colon-rettale tramite l’intelligenza artificiale rappresenta un avanzamento cruciale nella diagnostica medica odierna. Sfruttando tecniche di apprendimento supervisionato e semi-supervisionato insieme al deep learning nella visione artificiale, ARCADIA trasforma i risultati ecografici in diagnosi automatizzate.
Il progetto
Il nostro approccio si concentra sull’abilitare il modello a valutare inizialmente metriche e parametri chiave. I medici possono poi rivedere e perfezionare queste valutazioni secondo necessità, migliorando l’accuratezza dei rapporti. I dati validati vengono archiviati in un data lake, migliorando continuamente la qualità del modello attraverso un addestramento iterativo. Questa sinergia ottimizza i flussi di lavoro clinici, offrendo diagnosi e pianificazione dei trattamenti più rapidi.
La struttura
Sopra: Calcolo on-premises utilizzando modelli dalla libreria DL-CRT, impiegando Python per compiti ad alta intensità di calcolo con supporto CPU e GPU.
Al centro: Aggregazione basata su cloud di dataset CRT, utilizzando TPU e GPU per la modellazione DL ad alte prestazioni, garantendo ottimizzazione e scalabilità del modello.
Sotto: Un data lake collaborativo con immagini provenienti da tutto il mondo, standardizzando i metadati e le annotazioni delle immagini per l’addestramento del modello DL.
Segmentazione delle immagini
Prima del rilascio, è essenziale un addestramento rigoroso. Sviluppare un sistema di segmentazione robusto comporta superare sfide come la mancanza di dati etichettati. La nostra soluzione include un modello semi-supervisionato capace di apprendere da dataset parzialmente annotati, ottimizzando la segmentazione delle immagini 2D e 3D cruciali per diagnosi accurate.
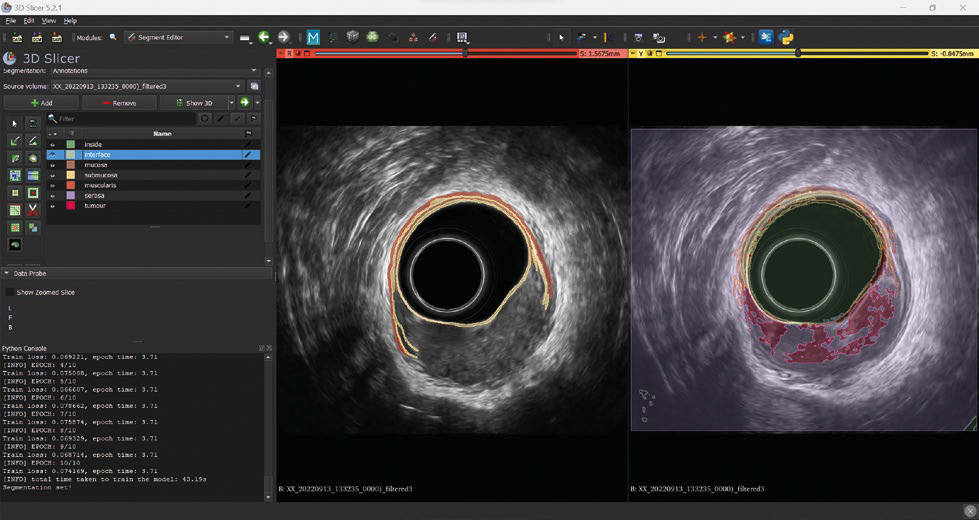
A sinistra, un’annotazione parziale su una singola slice, dove sono annotate solo le classi ‘interfaccia’, ‘mucosa’, ‘sottomucosa’ e ‘muscolaris’. A destra, la segmentazione suggerita dal modello durante la fase di addestramento.
Apprendimento Semi-Supervisionato
Addestrando con dati parzialmente etichettati, il nostro approccio massimizza l’efficienza riducendo i costi associati all’annotazione manuale. Questo metodo migliora la precisione del modello raffinando iterativamente la segmentazione attraverso la validazione degli esperti.
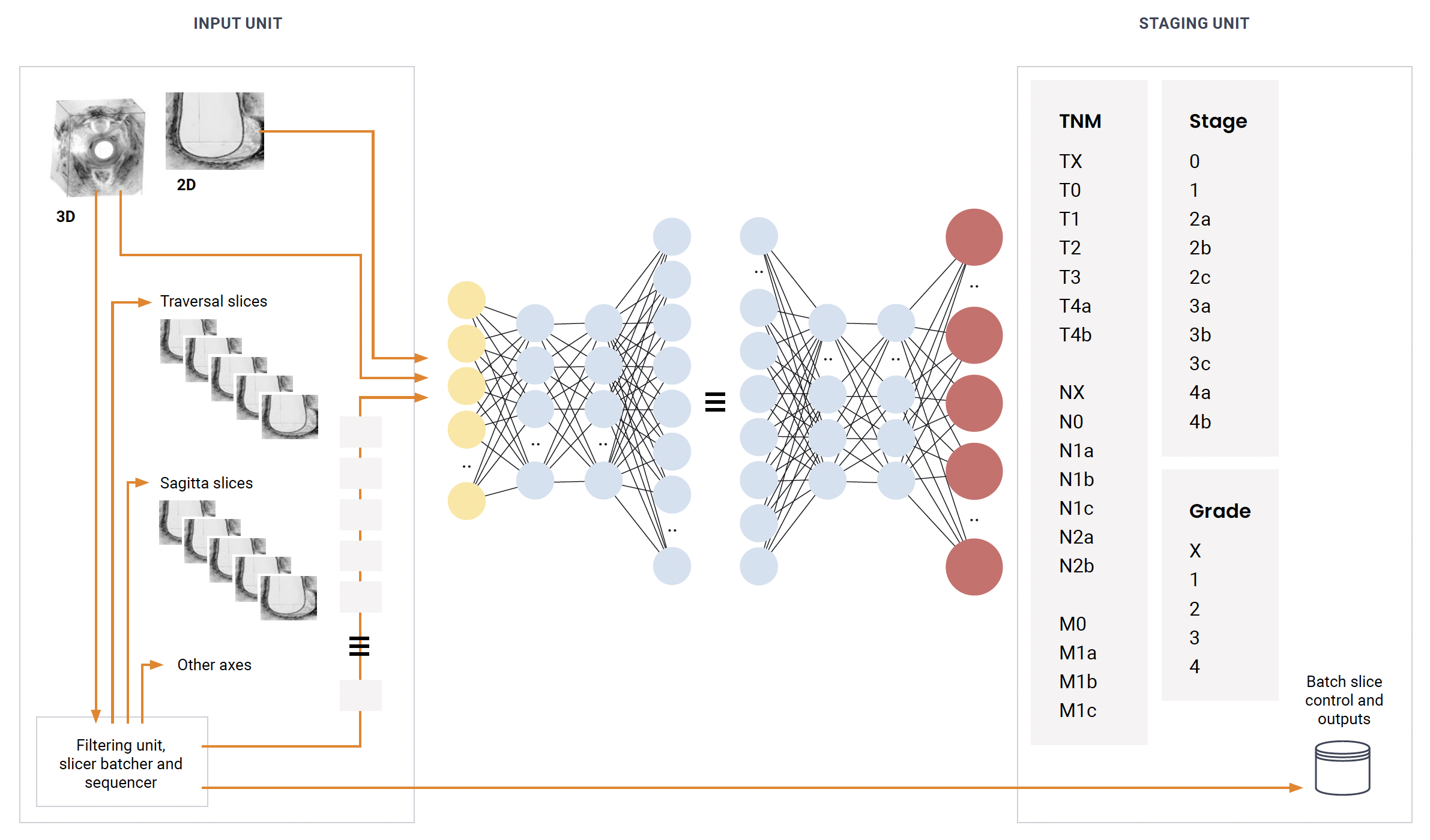
Il modello
Il cuore del nostro sistema è un modello Unet, un’architettura CNN adatta ai compiti di segmentazione delle immagini. Addestrato inizialmente su immagini completamente etichettate e integrato con dati parzialmente etichettati, estrae e raffina caratteristiche cruciali per l’identificazione e la stadiazione precisa dei tumori. La scalabilità del modello permette di estendere l’analisi da 2D a volumi 3D completi, culminando in mappe di segmentazione dettagliate essenziali per la decisione clinica.
